Coronavirus al microscopio elettronico - Dal sito della Scripps Uuniversity https://www.scripps.edu/news-and-events/press-room/2020/20200317-andersen-covid-19-coronavirus.html
20/03/2020
Coronavirus - Classificazione, struttura, epidemiologia, vie di trasmissione, manifestazioni cliniche- Una panoramica d'insieme
(Ultimo aggiornamento: 04/01/2021)
Introduzione
I coronavirus sono importanti patogeni per gli esseri umani e gli animali, essendo causa di infezioni respiratorie e intestinali. Scoperti negli anni 1960 [1,2], furono considerati relativamente innocui per l’uomo prima dell’identificazione del SARS-CoV come causa della severe acute respiratory syndrome (SARS), un’epidemia avvenuta in Cina nel biennio 2002-2003 [3,4]. Circa un decennio dopo, il Middle East respiratory syndrome coronavirus (MERS-CoV), un altro coronavirus patogeno, fu isolato in alcuni paesi mediorientali in circa 1800 soggetti durante un’epidemia di polmonite che aveva caratteristiche cliniche simili a quelle della SARS [5,6]. Alla fine del 2019, un nuovo coronavirus, il virus SARS-Cov 2 precedentemente designato 2019-nCoV [7], fu identificato come la causa di un focolaio di casi di polmonite a Wuhan, una città cinese della provincia di Hubei. Nei mesi successivi l’infezione si diffuse in altre zone della Cina e in almeno 70 altre nazioni, Italia compresa, diventando un'emergenza sanitaria globale, finché, nel marzo 2020, l’OMS fu costretta a dichiarare la pandemia. Durante le epidemie, i coronavirus sono la causa di oltre un terzo di tutte le infezioni delle vie respiratorie acquisite in comunità negli adulti; probabilmente svolgono anche un ruolo in alcune infezioni respiratorie gravi sia nei bambini che negli adulti [1]. Inoltre, l’apparato gastrointestinale è considerato un reservoir ed una possibile via di trasmissione di alcuni coronavirus [8], i quali sono stati chiamati in causa nell’eziopatogenesi di una minoranza di casi di diarrea nei neonati e nei bambini e dei sintomi gastrointestinali che si osservano in circa 1/3 dei pazienti nel corso delle epidemie da coronavirus [8,9]. Il loro ruolo in alcune malattie del sistema nervoso centrale nell’uomo, ad eccezione di qualche singolo caso in soggetti con grave immunodepressione, non è stato finora dimostrato. In questo articolo discutiamo la microbiologia, l'epidemiologia, le manifestazioni cliniche, la diagnosi, il trattamento e la prevenzione delle infezioni da coronavirus acquisite in comunità.
Non è stato defintivamente accertato se le infezioni da coronavirus umani siano trasmessi verticalmente dalla madre al feto durante la gravidanza. (vedere anche " Infezioni in gravidanza da coronavirus (COVID-19, SARS, MERS) – Vademecum per ginecologi ed ostetriche ")
Virologia
I coronavirus appartengono alla famiglia dei Coronaviridae, ordine dei Nidovirales, virus che si replicano utilizzando un set nidificato di mRNA [10,11]. La tassonomia di questi virus è cambiata più volte negli anni, ed è probabile che, con la scoperta di nuovi membri della famiglia, subisca ulteriori variazioni in futuro. I coronavirus erano stati inizialmente divisi in tre gruppi, a loro volta suddivisi in sottogruppi in base all'analisi sierologica e, oggi, a quella genetica. I coronavirus sono ulteriormente classificati in quattro generi: alfa, beta, gamma e delta coronavirus (vedi dopo, Classificazione dei coronavirus). I coronavirus umani (HCoVs) appartengono a due di questi generi: alfa coronavirus (HCoV-229E e HCoV-NL63) e beta coronavirus (che comprende HCoV-HKU1, HCoV-OC43, il coronavirus della sindrome respiratoria del Medio Oriente [MERS-CoV] e il SARS-CoV che causa la grave sindrome respiratoria acuta coronavirus o SARS) [1,3].
Struttura dei coronavirus
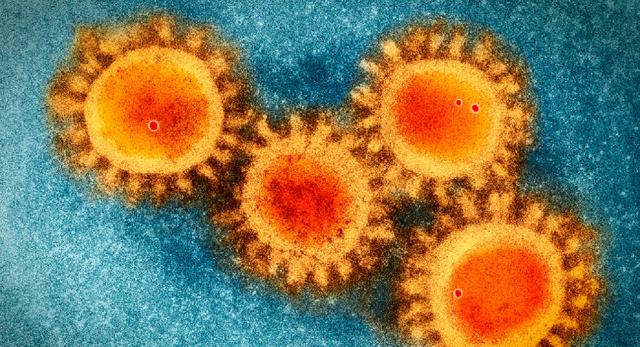
I coronavirus sono particelle sferiche con un diametro di circa 125 nm, il cui RNA è costituito da un monofilamento ad avvolgimento positivo, con un nucleocapside a simmetria elicoidale. Questa caratteristica è poco frequente nei virus a RNA ad avvolgimento positivo, mentre è più frequente nei virus a RNA con avvolgimento in senso negativo [11]. Il nome coronavirus deriva dal loro caratteristico aspetto nelle micrografie elettroniche che ricorda quello della corona solare (figura 1) [1,11]. Questi virus possiedono tra i più grandi genomi virali conosciuti, con una lunghezza compresa tra 27 e 32 kb. La membrana derivata dall'ospite è tempestata di punte o spine (spike), costituite dalla glicoproteina S (spike), che protrudono all’esterno della membrana stessa. Quest’ultima avvolge il genoma, che è a sua volta racchiuso in un nucleocapside, di forma elicoidale nella sua forma libera, ma che assume una forma approssimativamente sferica nella particella virale [12]. La replica dell'RNA virale avviene mediante un meccanismo caratteristico nel citoplasma della cellula ospite, dove l'RNA polimerasi si lega a una sequenza leader e quindi si stacca e riattacca in più posizioni, consentendo la produzione di un insieme annidato di molecole di mRNA con estremità 3' comuni [13]. HCoV-229E, HCoV-NL63 e il coronavirus SARS-Cov possiedono quattro geni che codificano rispettivamente per le proteine strutturali S, M, N ed E, mentre HCoV-OC43 e HCoV-HKU1 contengono anche un quinto gene che codifica per la proteina HE [11].
- La proteina S (spike) si proietta attraverso l'involucro virale e forma le caratteristiche “punte” o spine (spike) nella "corona" del coronavirus. La proteina S ha un peso molecolare di circa 150 kilodalton; è una proteina di fusione di classe 1 fortemente glicosilata; si associa in un omotrimero che permette il suo legame e la successiva fusione con i recettori di membrana della cellula ospite, rappresentati dall’enzima di conversione dell’angiotensina 2, angiotensin-converting enzyme 2 (ACE2) [11,14]. I principali determinanti antigenici che stimolano la comparsa di anticorpi neutralizzanti, nonché i più importanti epitopi bersaglio dei linfociti citotossici, sono localizzati sulla proteina S [15].
- La proteina M (di membrana o matrice) è la proteina strutturale più abbondante dei virioni, nei quali è presente come dimero. La proteina M ha un breve dominio N-terminale che si proietta sulla superficie esterna dell'involucro e lo circonda per tre volte, lasciando un lungo dominio terminale C all'interno dell'involucro stesso. La proteina M svolge un ruolo importante nell'assemblaggio delle particelle virali e nel mantenimento della loro forma, promuovendo la curvatura del virione e il legame con il nuclocapside [16,17].
- La proteina del nucleocapside (N) si associa al genoma dell'RNA per formare il nucleocapside all’interno del quale è la sola proteina presente [13]. La proteina N, oltre ad avere un ruolo nella regolazione della sintesi dell'RNA virale, probabilmente interagisce con la proteina M durante la gemmazione del virus [18,19]. È anche importante per l’ incapsidamento dell’RNA virale e agisce anche come antagonista dell’interferone; inoltre causa un’aumentata sintesi di una protrombinasi che svolgerebbe ruolo determinante nell’insorgenza di epatiti fulminanti in topi infettati da MHV-3 [20]. Sono stati identificati linfociti T citotossici che riconoscono alcuni epitopi della proteina N che potrebbero essere importanti per lo sviluppo di un vaccino [21].
- La glicoproteina (HE), emoagglutinina-esterasi, si trova solo in alcuni betacoronavirus, fra i quali HCoV-OC43 e HKU1. La porzione di proteina con funzione di emoagglutinina si lega all'acido neuraminico sulla superficie della cellula ospite, permettendo probabilmente l'assorbimento iniziale del virus sulla membrana e la sua diffusione sulle mucose delle vie respiratorie [22,23]. La porzione con attività esterasica scinde i gruppi acetilici dall'acido neuraminico. I geni HE dei coronavirus hanno un'omologia di sequenza con la glicoproteina HE del virus influenzale C e potrebbero essere il risultato di una ricombinazione tra i due virus [24,25]. Infatti, il pretrattamento degli eritrociti con il coronavirus bovino o il con virus dell'influenza C abolisce la capacità di agglutinare gli eritrociti sia dei coronavirus che del virus dell'influenza C, mentre il virus dell'influenza A conserva intatta la sua capacità di legare e agglutinare gli eritrociti [25]. La proteina HE probabilmente è importante per l’ingresso del virus nella cellula ospite e per la sua fuoriuscita dalla stessa, ma non è necessaria per la replicazione virale ( Lissenberg et al. 2005). La proteina E ha un PM fra 8 e 12 kD ed è presente in piccole quantità nell’envelope. In sua assenza, virus ricombinanti sembrano perdere parte della loro letalità, forse per difettoso assemblaggio del virione che annulla, completamente in alcuni coronavirus e parzialmente in altri, la capacità dei coronavirus di fuoruscire dalla cellula infettata [3,24,27]. La funzione della proteina E non è nota, sebbene, almeno nel SARS-CoV, la proteina E sia necessaria insieme alle proteine M e N per il corretto assemblaggio e rilascio del virus [22]. Inoltre sembra avere di attività di canale ionico, in quanto favorirebbe l’ingresso degli ioni calcio necessari per la crescita virale [24].
Oltre a queste proteine strutturali ce ne sono altre, codificate da un numero variabile di geni da uno a otto a seconda del ceppo virale; ognuno di questi geni “accessori” può codificare per più isoforme della stessa proteina. Le proteine accessorie non hanno alcuna omologia di struttura con altre proteine virali o cellulari e non sono necessarie per la replicazione virale in colture cellulari in vitro [28,29].
Classificazione dei coronavirus
I coronavirus animali e umani sono classificati in quattro generi distinti [2,3]. I coronavirus più importanti per le patologie che causano nell'uomo sono : HCoV-229E, HCoV-NL63, HCoV-OC43, HCoV-HKU1, SARS-CoV- MERS-CoV emerso nel 2012 e il nuovo SARS-CoV 2. Il genere alphacoronavirus comprende due specie virali umane, HCoV-229E e HCoV-NL63. L'HCoV-229E, come come numerosi alfacoronavirus animali, utilizza l'aminopeptidasi N (APN) (CD13) come recettore principale per penetrare nelle cellule umane [30]. Al contrario, HCoV-NL63 e il SARS-CoV (un betacoronavirus), utilizzano l'enzima-di conversione dell'angiotensina 2 (ACE-2) come recettore [31]. Due delle specie umane non SARS appartengono al genere betacoronavirus, assieme a HCoV-OC43 e HCoV-HKU1, a diversi virus dei pipistrelli, a MERS-CoV e SARS-CoV [32,33], sebbene gli ultimi due siano geneticamente alquanto distanti da HCoV-OC43 e HCoV-HKU1. Importanti betacoronavirus animali sono il virus dell'epatite del topo, un modello di laboratorio sia per l'epatite virale che per la malattia demielinizzante del sistema nervoso centrale, e il coronavirus bovino. Il coronavirus bovino è così simile all'HCoV-OC43 che i due virus sono stati fusi in una singola specie chiamata betacoronavirus 1 [34]. Si ritiene che HCoV-OC43 sia passato da un ospite animale all’uomo fin dal 1890 [35]. Il genere gammacoronavirus contiene principalmente coronavirus aviari, il più importante dei quali è il virus della bronchite infettiva dei polli, un importante patogeno veterinario che causa malattie del tratto respiratorio e riproduttivo nei polli. Il genere deltacoronavirus contiene coronavirus aviari scoperti di recente e trovati in diverse specie di uccelli canori.
In precedenza i coronavirus erano classificati in tre gruppi e in vari sottogruppi in base all’analisi del loro genoma [3]. Per quanto riguarda i coronavirus più importanti per l’uomo HCoV-229E e HCoV-NL63 erano stati posti nel gruppo 1b, HCoV-OC43 e HCoV-HKU nel gruppo 2a, il SARS-COV nel gruppo 2b [3].
Una lista, probabilmente destinata ad allungarsi in futuro, di coronavirus patogeni per l’uomo è la seguente [36]:
- Human Coronavirus 229E (HCoV-229E)
- Human Coronavirus OC43 (HCoV-OC43)
- Human Coronavirus NL63 (HCoV-NL63)
- Human Coronavirus HKU1 (HCoV-HFU1[3])
- Severe Acute Respiratory Syndrome Coronavirus (SARS-CoV)
- Sindrome respiratoria mediorientale da Coronavirus (MERS-CoV), conosciuto anche come Novel Coronavirus 2012 (2012-nCoV)
- Human Coronavirus Erasmus Medical Center/2012 HCoV-EMC/2012
- Severe Acute Respiratory Syndrome Coronavirus-2 (SARS-CoV-2), responsabile della malattia COVID-19.
Adesione, penetrazione e replicazione dei coronavirus
Tutti i coronavirus adottano la stessa strategia di base per la loro azione. L'iniziale adesione del virus alla cellula ospite prevede l’interazione fra la proteina S e il suo recettore sulla membrana cellulare. Molti coronavirus alfa utilizzano l'aminopeptidasi N come recettore; SARS-CoV, HCoV-NL63 e il nuovo SARS-CoV 2, usano ACE-2; MHV penetra attraverso CEACAM1, mentre MERS-CoV si lega alla dipeptidil-dipeptidasi 4 (DPP4 o CD26) [13]. Dopo l’adesione avviene la fusione della membrana virale con quella cellulare, cui segue la traduzione del genoma. Viene assemblato quindi il complesso replicasi-trascrittasi che crea l’ambiente favorevole alla replicazione dell’RNA virale; gli RNA delle proteine vengono tradotti e trasportati sul reticolo endoplasmatico cellulare e nelll’apparato di Golgi dove, sotto la guida della proteina M si svolgono le interazioni necessarie per l’assemblaggio dei coronavirus [13]. La replicazione virale avviene probabilmente sulle vescicole a doppia membrana (double membrane vesicles, DMV) formatesi a carico delle cisterne del reticolo endoplasmatico della cellula ospite [37]. I nuovi virioni così formatisi verranno poi estrusi dalla cellula ospite [3,38].
Patogenicità dei coronavirus
Prima dell’epidemia di SARS nel 2002-2003, soltanto due coronavirus umani, HCoV-OC43 and HCoV-229E, erano considerati patogeni importanti per l’uomo in quanto erano stati isolati da pazienti con infezioni delle vie aeree superiori, nei quali, occasionalmente, provocavano anche gravi malattie polmonari, soprattutto in soggetti anziani, in neonati o nei pazienti immunodepressi [39]. Al contrario degli altri due coronavirus sopra ricordati, SARS-COV causa una grave sindrome respiratoria acuta, con quasi il 10% di letalità osservata nel 2002-2003 con punte fino a 50% nei soggetti anziani [4]. A differenza di HCoV-OC43 and HCoV-229E, che causano infezioni delle vie respiratorie, SARS -CoV provocò anche malattie sistemiche, con segni di infezioni a carico dell’apparato gastrointestinale, del fegato, dei reni e del sistema nervoso e, meno frequentemente, di altri organi e tessuti [40].
Nessuno dei coronavirus umani acquisiti in comunità (HCoV-OC43, HCoV-NE63, HCoV-HKU1 e HCoV-229E, SARS-CoV,) si replica facilmente nelle colture di tessuti e, fino a poco tempo fa, questa loro proprietà ha rappresentato un grosso ostacolo per i ricercatori. Sia HCoV-229E che HCoV-OC43 furono scoperti negli anni '60 e in esperimenti su volontari furono identificati come causa dei comuni raffreddori negli adulti [13,24]. Studi condotti negli anni '70 e '80 li associarono a circa un terzo delle infezioni del tratto respiratorio superiore durante le epidemie invernali, al 5- 15% delle sindromi da raffreddamento negli adulti e ad una parte delle malattie delle vie respiratorie inferiori nei bambini [9,41].
In seguito, pochi furono gli studi condotti sui coronavirus fino all'emergere della SARS nel 2002. Lo sviluppo e diffusione di metodi diagnostici molecolari ha favorito un ritorno di interesse dei ricercatori verso i coronavirus. Ciò ha favorito la rapida identificazione di HCoV-NL63 e HCoV-HKU1 e la scoperta della loro distribuzione su scala mondiale [9,9,13,41]. La reazione polimerasi a catena (PCR) è utilizzata per la diagnosi rapida dell’infezione dei coronavirus umani e per gli studi epidemiologi e di patogenicità.
I coronavirus causano diverse patologie negli animali, sia nel bestiame che negli animali domestici o da compagnia come maiali, mucche, polli, cani e gatti; ciò ha stimolato numerose ricerche su questi virus nell'ultima metà del XX secolo [3]. Per esempio, il virus della gastroenterite trasmissibile (TGEV) e il virus della diarrea epidemica dei porcellini (PEDV) causano una grave gastroenterite nei giovani maialini. La significativa morbilità e mortalità che ne conseguono sono all’origine di notevoli perdite economiche. Il virus emagglutinante dell’encefalomielite porcina (PHEV) causa per lo più infezione enterica, ma ha la capacità di infettare il sistema nervoso, causando encefalite, diarrea e vomito nei maiali. Il coronavirus enterico felino (FCoV) provoca un'infezione lieve o asintomatica nei gatti domestici, ma durante l'infezione latente, la mutazione trasforma il virus in un ceppo altamente virulento di FCoV (Feline Infectious Peritonitis Virus, FIPV), che causa una malattia letale chiamata peritonite infettiva felina (FIP). La FIP ha diverse forme, alcune delle quali somigliano molto ad una malattia umana, la sarcoidosi. FIPV ha un notevole tropismo per i macrofagi e si ritiene che provochi un esagerata sintesi di citochine e/o chemochine con deplezione linfocitaria e conseguente malattia letale [3,13]. CoV bovino, CoV dei ratti e il virus della bronchite infettiva (IBV) causano sindromi di varia gravità, con infezioni da lievi a letali delle vie respiratorie nei bovini, nei ratti e nei polli, rispettivamente. Il CoV bovino provoca perdite economiche nell'industria del bestiame e si è anche adattato in diversi ruminanti, tra cui alci, cervi e cammelli. Oltre a gravi malattie respiratorie, il virus provoca perdita di peso, disidratazione, diminuzione della produzione di latte e depressione [3,13]. Alcuni ceppi di IBV colpiscono anche il tratto uro-genitale dei polli causando malattie renali. L'IBV riduce significativamente la produzione di uova e rallenta l’aumento di peso del pollame, causando ogni anno notevoli perdite negli allevatori. Più recentemente, un nuovo coronavirus di nome SW1 è stato identificato in una balena Beluga deceduta in un acquario degli USA [42]. Nel fegato della balena all’autopsia furono identificate un gran numero di particelle virali in grado di causare malattie respiratorie e insufficienza epatica acuta in altri animali di laboratorio. Il sequenziamento del tessuto epatico permise di identificare chiaramente il virus come un coronavirus.
I ricercatori hanno manifestato negli ultimi anni un forte interesse nell’identificazione nuovi CoV nei pipistrelli, dal momento che questi sono gli ospiti finali più probabili per SARS-CoV e MERS-CoV, e le centinaia di nuovi coronavirus che sono stati identificati nell'ultimo decennio [43]. I pipistrelli sono pertanto emersi come il reservoir animale che ospita il maggior numero di genotipi di coronavirus [44].
(Vedere " COVID-19 e coinfezioni batteriche, virali e fungine" e "COVID-19 e ipercoagulabilità : la triade di Virchow è sempre attuale ")
Trasmissione fra specie diverse (salto di specie) dei coronavirus
Una caratteristica sorprendente dell'epidemia di SARS del 2002-2003 è stata la capacità del SARS-CoV di effettuare il “salto di specie” passando dalla civetta mascherata delle palme himalayana (Pagkuma larvata) o zibetto, dal cane procione (Nyctereutes procyonoides) e dal tasso furetto cinese (Melogale moschata) all’uomo (Guan et al. 2003)(Figura 3A). La facilità di saltare sulle specie animali superiori spiega anche il grande numero di specie animali, uomo compreso, infettati da virus strettamente correlati al coronavirus bovino. Nel caso della SARS, la trasmissione avvenne nei mercati della vendita al dettaglio di animali vivi con il passaggio dell’infezione ai venditori di animali. Sembra che varianti del SARS-CoV legate al ceppo epidemico infettino abbastanza frequentemente le popolazioni umane nei mercati di animali vivi del sud est asiatico, come dimostrato dall'elevato tasso di sieropositività rilevato negli addestratori di animali, anche in casi asintomatici che non hanno sviluppato una malattia simile alla SARS. In quell'epidemia il caso indice fu rappresentato da un medico curante degli addetti a quei mercati che, dopo essere stato infettato, trasmise a sua volta l’infezione a numerosi contatti (Perlman e Netland 2009; Riley et al. 2003).
Le analisi genetiche del virus isolate dallo zibetto e dagli esseri umani durante l'epidemia hanno dimostrato che il virus ha subito un rapido adattamento nelle varie specie [45,46], con mutazioni e ricombinazioni principalmente nel dominio di legame del recettore della proteina S (RBD, receptor binding domain) che hanno aumentato l’efficienza della proteina S di infettare le cellule umane [47]; in particolare le mutazioni K479N e S487T nell'RBD sono state fondamentali per il legame della proteina S al recettore umano (hACE2). Questi risultati sono stati recentemente confermati utilizzando linee cellulari che esprimono ACE2 dello zibetto o dell’uomo (hACE2) [48].
Diverse osservazioni depongono contro l’ipotesi che lo zibetto e altri animali selvatici venduti nei mercati fossero i principali serbatoi del virus. Dal momento che CoV SARS-like sono stati isolati dai pipistrelli a ferro di cavallo cinesi [49,50], che erano anch’essi venduti negli stessi mercati, il virus potrebbe essersi recentemente diffuso dai pipistrelli ad altri mammiferi come le civette delle palme e poi agli esseri umani (Figura 3A). D’accordo con l’ipotesi di una recente diffusione, sono stati rilevati anticorpi contro la SARS-CoV a livelli estremamente bassi (0,008%) in studi di popolazione a Hong Kong [51]. Oltre al SARS-CoV, ci sono altri esempi di trasmissione di coronavirus tra specie diverse, in alcuni casi risalenti a centinaia di anni fa [3].
Epidemiologia dei coronavirus
Nelle regioni con clima temperato, le infezioni respiratorie da coronavirus si verificano principalmente in inverno, anche se si osservano anche picchi più piccoli in autunno o in primavera e le infezioni possono verificarsi in qualsiasi momento dell'anno [52]. Negli Stati Uniti il picco dei testi positivi si osserva ogni anno fra Dicembre e Marzo [52]. Nei bambini ricoverati a Guangzhou, in Cina, una regione subtropicale, sono stati focolai in quasi ogni periodo dell'anno, con una prevalenza in primavera e in autunno [53]. In alcuni studi , HCoV-OC43, HCoV-NL63, HCoV-229E e HCoV-HKU1 predominano in modo imprevedibile in alcuni anni e in alcune parti del mondo [[53,54,54,55]. In quasi tutte questi studi, HCoV-OC43 è il più comune dei quattro ceppi, seguito da HCoV-NL63, ma la prevalenza dei vari ceppi in un determinato anno non è facilmente prevedibile [21,56,57]. In Tailandia, il picco si osserva in genere fra Maggio e Ottobre, i mesi caratterizzati da maggiore piovosità [58]. In Scozia, nell’ambito di un grosso studio sulla diffusione dei virus in adulti e bambini con malattie respiratorie acute, furono effettuati prelievi per 11 gruppi di virus in oltre 44.000 episodi in nove anni, nel periodo compreso fra il 2005 e 2013, compresa anche la pandemia di influenza A del 2009 [59]. Le infezioni da HCoV erano più frequenti in inverno, erano distribuite in tutte le fasce d'età ed erano meno frequenti di quelle causate da rinovirus, influenza o virus respiratorio sinciziale ma più comuni di altri virus respiratori; le coinfezioni erano relativamente comuni, in particolare nei bambini piccoli [59].
Nello studio norvegese prima citato [54], durato nove anni nel quale furono arruolati tutti i bambini di età inferiore a 16 anni ricoverati in ospedale per malattia respiratoria acuta, fu scoperto che HCoV-OC43 e HCoV-NL63 erano i virus più frequenti e diventavano epidemici ogni due inverni, che HCoV-HKU-1 di solito prevaleva anch’esso ogni due inverni, ma negli anni in cui HCoV-OC43 e HCoV-NL63 non lo facevano e che la presenza di 229E era insolita [54].
Vie di trasmissione dei coronavirus
I coronavirus respiratori probabilmente si diffondono in modo simile a quello dei rinovirus, attraverso il contatto diretto con secrezioni infette o le goccioline di aerosol di maggior diametro. L'immunità si sviluppa subito dopo l'infezione ma diminuisce gradualmente nel tempo. La reinfezione è frequente, forse a causa dell'immunità calante o, più probabilmente, a causa della variazione antigenica all'interno delle specie [60]. In ambito ospedaliero, la diffusione tra i pazienti pediatrici ricoverati nei reparti di terapia intensiva neonatale si verifica probabilmente attraverso lo staff e i parenti infetti [61]. I focolai sono frequenti nelle strutture residenziali e di assistenza per anziani, nei quali i coronavirus sono fra i virus più frequentemente isolati, quasi sempre in soggetti portatori di diversi patogeni virali nelle loro vie respiratorie [62]. Anche se il contagio avviene principalmente attraverso le goccioline respiratorie, le infezioni del tratto gastrointestinale e, forse delle vie urinarie, potrebbe facilitare la diffusione di alcuni coronavirus per altre vie [3]. (Vedere anche " Covid-19 . Trasmissione con le acque reflue e conseguenze sul contagio per via oro-fecale")
Manifestazioni cliniche
I coronavirus umani causano essenzialmente una percentuale stimata attorno al 15% delle sindromi da raffreddamento in periodo invernale, raramente gravi sindromi respiratorie acute. Meno frequenti sono le sindromi gastroenteriche, mentre non esistono prove certe di un loro ruolo eziopatogenetico in altre malattie umane
Apparato respiratorio
HCoV-229E e HCoV-OC43 hanno dimostrato di essere patogeni nell'uomo in studi sui volontari nei quali, insieme ad altri ceppi di coronavirus meno noti, hanno riprodotto raffreddori molto simili a quelli indotti da rinovirus e tipicamente caratterizzati da congestione nasale e rinorrea e altri sintomi a carico del tratto respiratorio superiore [56,62,63]. Si ipotizza che HCoV-NL63 e HCoV-HKU1 abbiano simile patogenicità, ma mancano prove a sostegno di tale ipotesi. Inoltre, i coronavirus sono dimostrabili periodicamente con la PCR in individui asintomatici di tutte le età [41,64]. HCoV-NL63 fu inizialmente isolato da bambini ricoverati in ospedale per gravi malattie respiratorie [65,66], mentre HCoV-HKU1 fu per la prima volta identificato in persone anziane con comorbilità [66,67,67]. È probabile che HCoV-NL63 abbia infettato nel corso dei secoli milioni di esseri umani, essendo stato stimato che la divergenza dal HCoV-229E sia avvenuta oltre un migliaio di anni fa [68].
I coronavirus umani probabilmente rappresentano il 5-10% di tutte le infezioni acute del tratto respiratorio superiore negli adulti [1], con focolai durante i quali il 25-35% delle infezioni respiratorie può essere attribuito a una singola specie. Come i rinovirus, i coronavirus colonizzano l'orecchio medio e sono stati implicati come importanti cause virali dell'otite media acuta nei bambini [69,70]. Studi sulle infezioni del tratto respiratorio che hanno incluso neonati e bambini asintomatici indicano che i coronavirus, come i rinovirus, sono spesso coinfezioni con altri virus respiratori e si trovano spesso in assenza di sintomi respiratori, suggerendo che, sebbene frequenti, la loro patogenicità in neonati e bambini sani possa essere relativamente bassa [69,71].
I coronavirus causano anche malattie respiratorie più gravi. Negli adulti con polmonite acquisita in comunità, i coronavirus vengono rilevati mediante PCR con frequenze simili o leggermente inferiori a quelle di altri virus respiratori come il virus dell'influenza, il rinovirus e il virus respiratorio sinciziale [1,64]. Il loro ruolo eziologico non è chiaro, in parte perché si trovano spesso copatogeni. In alcuni studi, è stato effettuato il campionamento simultaneo di adulti sani. In diversi studi, i coronavirus sono stati dimostrati più frequentemente nei soggetti con polmonite o altre infezioni delle vie respiratorie inferiori rispetto ai soggetti sani [57,72–74]. Il rapporto tra infezioni ambulatoriali da HCoV-OC43 e infezioni ospedaliere è triplicato rispetto a quello per HCoV-229E, suggerendo che HCoV-OC43 potrebbe avere un maggiore impatto clinico. Un altro studio sulle infezioni respiratorie acute gravi dal 2010 al 2014 in Arizona, USA, il virus dell'influenza risultò il patogeno più frequente, con 49 (16%) su 309 casi, seguito dal metapneumovirus umano (25 casi, 8%), dai virus parainfluenzali (20 casi,6,5%). dai coronavirus (16 casi, 5%,) e dal virus respiratorio sinciziale (11 casi); tra i coronavirus, predominava HCo, V-OC43 [75]. I coronavirus sono importanti cause di sindromi simil-influenzali, di bronchite cronica e polmonite negli anziani, nei quali la loro frequenza è inferiore a quella dell'influenza e del virus respiratorio sinciziale ma simile a quella dei rinovirus [72,75–78]. Sono stati descritti diversi focolai di malattia respiratoria HCoV-OC43 negli anziani che vivono in resistenze sanitarie assistite [76,79], con tassi di mortalità dello dell'8%. È stato anche riportato un caso fatale di sindrome da distress respiratorio acuto in una donna israeliana di 76 anni senza patologie di base e monoinfezione con HCoV-NL63 [80].
Nei bambini ricoverati con infezione da HCoV e malattie respiratorie, la maggioranza ha meno di cinque anni e presenta spesso comorbilità come malattie cardiache, malattie polmonari croniche o malformazioni congenite [71].
I coronavirus sono stati dimostrati nel 4-6 percento degli adulti durante riacutizzazione della broncopneumopatia cronica ostruttiva (meno frequentemente dei rinovirus e del virus respiratorio sinciziale ma con frequenze simili al virus influenzale e più frequentemente dei virus della parainfluenza, del metapneumovirus umano e degli adenovirus) [64]. I Coronavirus sono stati anche associati a crisi acute di asma sia nei bambini che negli adulti [81,82]. Sono presenti con frequenze variabili, che vanno dal 2 all'8%, in neonati e bambini piccoli ricoverati in ospedale con polmonite acquisita in comunità, e sono stati identificati ancora più frequentemente in pazienti ambulatoriali con malattie delle vie respiratorie [3]. Sono anche una causa importante di infezioni nosocomiali nelle unità di terapia intensiva neonatale [83]. Uno dei coronavirus umani scoperti più di recente, HCoV-NL63, è stato associato alla laringotrcheobronchite acuta spastica (croup) nei bambini [84].
I coronavirus causano polmonite negli ospiti immunocompromessi [85] sia per cause congenite [86] che per neoplasie ematologiche [87,88] o per infezione da HIV [89,90]. I campioni di liquido di lavaggio broncoalveolare di ventotto pazienti sottoposti a trapianto di cellule ematopoietiche (HCT) infetti da HCoV furono confrontati con quelli prelevati a soggetti con infezioni da virus influenzale, RSV e virus parainfluenzale seguiti nello stesso centro [88]. All’analisi multivariata, non furono osservate significative differenze di sopravvivenza tra i pazienti con infezione da HCoV e quelli infettati da altri virus respiratori. Kumar et al [91] hanno osservato un'associazione tra infezione da coronavirus, anche asintomatica, e rigetto acuto del trapianto, oltre che con la sindrome da bronchiolite obliterante in pazienti sottoposti a trapianto di polmone, sebbene l'associazione fosse meno forte rispetto ad altri virus respiratori.
Apparato gastro-enterico
L’ipotesi che i coronavirus producano diarrea nell'uomo è interessante a causa della loro chiara patogenicità intestinale negli animali [13]. I primi studi sull'uomo si basavano sulla ricerca di "particelle simili al coronavirus" (CVLP) mediante microscopia elettronica nei campioni di feci. Gli studi più convincenti hanno mostrato una forte associazione tra la presenza di CVLP e la diarrea nei neonati [1,13,92] o l'enterocolite necrotizzante nei neonati [92]. Gerna et al [93] hanno purificato CVLP che sembrano essere antigenicamente correlati a HCoV-OC43.
Tutte e quattro le specie di HCoV sono state trovate mediante RT-PCR nelle feci e nei tamponi orofaringei di una piccola percentuale di neonati e bambini ricoverati in ospedale con diarrea e/o sintomi respiratori [94]. Tuttavia, una percentuale non trascurabile di bambini ricoverati nello stesso ospedale per altri motivi o asintomatici avevano un tampone positivo per coronavirus, per cui non è facile sostenere un ruolo patogenetico dei coronavirus nella sintomatologia intestinale [94]. Risultati simili furono ottenuti in un altro studio, nel quale tutte e quattro le specie di coronavirus furono trovate nelle feci del 2,5% di 878 bambini con diarrea e del 2% circa di 112 bambini asintomatici mediante RT-PCR [95]. Bisogna tuttavia precisare che la maggior parte dei campioni fecali positivi al coronavirus nei bambini con diarrea contenevano anche altri patogeni noti, come il rotavirus o il norovirus [94,95]. CoV-HKU1 fu identificato nelle feci in 4 (0,8 %) su 479 pazienti di età pediatrica o adulti con malattie gastrointestinali; le altre specie di HCoV cercate risultarono assenti [96].L'associazione tra manifestazioni gastrointestinali (diarrea, vomito, nausea,dolore addominale), sintomi respiratori e sintomi sistemici (febbre, brividi, mal di testa o mialgia) fu analizzata in soggetti adulti seguiti in ambulatorio dal proprio medico di medicina generale [97]. Gli autori dello studio cercarono gruppi di virus nei campioni respiratori e nelle feci e vari batteri solo nelle feci. I sintomi gastrointestinali, che erano presenti nel 57% dei pazienti, si manifestavano con maggiore frequenza in soggetti con febbre> 39 ° C mal di testa, infettati da patogeni gastrointestinali o con infezione respiratoria da HCoV. Sebbene alcuni campioni di feci fossero positivi per HCoV, gli autori ipotizzarono che questi fossero con ogni probabilità virus provenienti dall’orofaringe con la deglutizione [97].
Malattie neurologiche
Numerosi coronavirus animali sono stati associati a malattie neurologiche croniche, stimolando la ricerca di un simile ruolo patogenetico dei coronavirus umani. I coronavirus umani acquisiti in comunità (HCoV) possono infettare diverse linee di cellule neurali in vitro [98]. Topi di tre settimane sviluppano un’encefalite vacuolizzante generalizzata dopo inoculazione intracerebrale con HCoV-OC43 [99]. Sequenze di RNA di HCoV-OC43 sono state rilevate nel liquido cerebrospinale di un ragazzo di 15 anni con una presunta encefalomielite acuta demielinizzante [100]. In un altro studio, l’intero genoma del HCoV-OC43 fu dimostrato all'autopsia in tutte le regioni cerebrali in un ragazzo di 11 mesi con immunodeficienza combinata grave ed encefalite acuta dopo trapianto di sangue del cordone ombelicale [101]. Ratti e topi infettati con alcuni ceppi del virus dell'epatite di topo (MHV) hanno sviluppato una grave encefalite demielinizzante simile alla sclerosi multipla (SM) [102]. Gli studi finora compiuti non hanno però fornito risultati univoci sull’eventuale associazione fra coronavirus e sclerosi multipla nell’uomo. È stato dimostrato che cloni di cellule T di pazienti con SM reagiscono sia con gli antigeni HCoV-229E che con le proteine basiche della mielina, suggerendo il mimetismo molecolare come base della comune patogenesi [103]. Alcuni ricercatori hanno identificato mediante PCR-RT l'RNA di due coronavirus umani, HCoV-OC43 e HCoV-229E, con una frequenza maggiore nel tessuto cerebrale del pazienti con SM rispetto agli individui sani; tuttavia. non fu possibile distinguere fra infezione opportunistica o silente e infezione patogeneticamente attiva nei casi di SM o di altre malattie neurodegenerative [104]. Nonostante i risultati di alcuni studi inducano ad ipotizzare una connessione eziologica tra coronavirus e SM o altre malattie demielinizzanti, l’ipotesi necessita ancora di dimostrazione definitiva [105].
Malattia di Kawasaki
L’associazione dell’infezione da coronavirus con la malattia di Kawasaki è stata suggerita da un gruppo di ricercatori e ha stimolato l’interesse di numerosi gruppi in tutto il mondo [106]. I risultati degli studi finora compiuti, comunque, suggeriscono che i coronavirus noti non abbiano alcun ruolo in questa malattia [107,108]. (Vedere "COVID-19 e complicanze cardiovascolari ")
Diagnosi
Poiché non esiste un trattamento efficace per le infezioni da coronavirus, arrivare ad una diagnosi precisa è di utilità limitata nei pazienti con sospetta infezione da coronavirus acquisita in comunità. Al contrario, la diagnosi del coronavirus 2019 (COVID-19), della sindrome respiratoria del Medio Oriente da coronavirus (MERS-CoV) e della sindrome respiratoria acuta grave da coronavirus (SARS-CoV) è di fondamentale importanza per comprendere la portata delle epidemie e limitarne la diffusione [12,109,110].
Oggi disponiamo di tecniche di laboratorio rapide che possono essere utilizzate per rilevare i coronavirus dai campioni rinofaringei basate sulla RT-PCR e sull’immunofluorescenza [111,112]. La RT-PCR è la metodica preferita grazie alla sua capacità di rilevare contemporaneamente tutti e quattro i ceppi di coronavirus umani più importanti per il loro ruolo causale nelle infezioni acquisite in comunità [111–113]. I coronavirus acquisiti in comunità sono difficili da replicare nella colture tessutali.
TRATTAMENTO E PREVENZIONE
Attualmente non esiste alcuna terapia efficace per le infezioni da coronavirus. Diversi antivirali e altri farmaci sono stati utilizzati durante l'epidemia di coronavirus della sindrome respiratoria acuta grave (SARS-CoV), ma l'efficacia di questi farmaci non è stata stabilita. La clorochina, che ha una potente attività antivirale contro il SARS-CoV [114] ha dimostrato di avere un'attività simile contro HCoV-229E in cellule in coltura [114] e contro HCoV-OC43 sia in cellule in coltura che in un modello murino [115]. Sono in corso studi sull'efficacia nell'uomo [1,12,116].
La prevenzione richiede l’adozione delle stesse precauzioni efficaci in altre infezioni virali, per esempio delle infezioni da rinovirus, che consistono nel lavaggio delle mani e nel corretto smaltimento dei materiali infettati da secrezioni nasali. Anche l'uso di disinfettanti di superficie è un momento importante nel controllo delle infezioni, poiché i coronavirus sembrano sopravvivere per uno o più giorni dopo essiccamento su superfici come acciaio inossidabile, plastica o tessuto [117].
L'efficacia di vari disinfettanti è stata esaminata sia sui virus in sospensione liquida sia sui virus essiccati su varie superfici [118]. Sono stati studiati i coronavirus umani, inclusi CoV-229E e SARS-CoV, nonché diversi coronavirus animali (ad esempio, virus dell'epatite di topo e virus della gastroenterite trasmissibile dei suini). Questi virus (sia in sospensione che essiccati sulle superfici) erano molto sensibili al 70% di etanolo, con riduzione della vitalità di oltre 3 log in pochi secondi [119–121]. Allo stesso modo, l'esaclorofene [122], soluzioni al 2% di glutaraldeide [119] o all’1% di povidone-iodio [120–122] hanno prodotto tutte percentuali soddisfacenti di riduzione della carica virale. La suscettibilità dei coronavirus al 6% di soluzioni di ipoclorito di sodio (l'agente attivo nella candeggina) è risultata molto variabile, ma un’eliminazione virale soddisfacente è stata raggiunta con concentrazioni di 1:40 o superiori [120,121]. I coronavirus sembrano resistenti al benzalconio cloruro e alla clorexidina a meno che non sia aggiunto etanolo al 70% [119].
Finora, c’è stato per diversi motivi scarso interesse per lo sviluppo di vaccini per i coronavirus non SARS acquisiti in comunità. Innanzitutto, sono state descritte quattro specie distinte e per almeno una di esse vi sono prove di variazione antigenica clinicamente significativa [12]. Inoltre, è stato dimostrato un peggioramento da parte del vaccino della malattia per un coronavirus animale, il coronavirus felino; l'ipersensibilità è stata indotta in alcuni animali dalla precedente esposizione a un vaccino contenente la proteina S, con la produzione di una malattia grave immunologicamente mediata, la peritonite infettiva felina, dopo reinfezione con un coronavirus [123]. Diversi ricercatori sono impegnati nello sviluppo di un vaccino contro i coronavirus più virulenti, SARS-CoV, MERS-CoV e il più recente COVID-19.
Informazioni sulla pandemia da COVID-19, sulle sue manifestazioni cliniche, la diagnosi, la prevenzione e la terapia di questa nuove sindrome sono fornite in altri articoli.
Riferimenti bibliografici
-
Kahn JS, McIntosh K. History and Recent Advances in Coronavirus Discovery. Pediatr Infect Dis J [Internet]. 2005 [cited 2020 Mar 18]; 24: S223. Available from: https://journals.lww.com/pidj/Fulltext/2005/11001/History_and_Recent_Advances_in_Coronavirus.12.aspx
-
Luk HKH, Li X, Fung J, Lau SKP, Woo PCY. Molecular epidemiology, evolution and phylogeny of SARS coronavirus. Infect Genet Evol J Mol Epidemiol Evol Genet Infect Dis. 2019; 71: 21–30.
-
Perlman S, Netland J. Coronaviruses post-SARS: Update on replication and pathogenesis. Nat Rev Microbiol [Internet]. 2009 [cited 2020 Mar 18]; 7: 439–50. Available from: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2830095/
-
Peiris JSM, Guan Y, Yuen KY. Severe acute respiratory syndrome. Nat Med. 2004; 10: S88-97.
-
Hajjar SA, Memish ZA, McIntosh K. Middle East Respiratory Syndrome Coronavirus (MERS-CoV): a perpetual challenge. Ann Saudi Med. 2013; 33: 427–36.
-
de Groot RJ, Baker SC, Baric RS, Brown CS, Drosten C, Enjuanes L, Fouchier RAM, Galiano M, Gorbalenya AE, Memish ZA, Perlman S, Poon LLM, Snijder EJ, et al. Middle East Respiratory Syndrome Coronavirus (MERS-CoV): Announcement of the Coronavirus Study Group. J Virol [Internet]. 2013 [cited 2020 Mar 18]; 87: 7790–2. Available from: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3700179/
-
WHO Director-General’s remarks at the media briefing on 2019-nCoV on 11 February 2020 [Internet]. [cited 2020 Mar 18]. Available from: https://www.who.int/dg/speeches/detail/who-director-general-s-remarks-at-the-media-briefing-on-2019-ncov-on-11-february-2020
-
Zhou J, Li C, Zhao G, Chu H, Wang D, Yan HH-N, Poon VK-M, Wen L, Wong BH-Y, Zhao X, Chiu MC, Yang D, Wang Y, et al. Human intestinal tract serves as an alternative infection route for Middle East respiratory syndrome coronavirus. Sci Adv. 2017; 3: eaao4966.
-
Chan JFW, Lau SKP, To KKW, Cheng VCC, Woo PCY, Yuen K-Y. Middle East respiratory syndrome coronavirus: another zoonotic betacoronavirus causing SARS-like disease. Clin Microbiol Rev. 2015; 28: 465–522.
-
Enjuanes L, Almazán F, Sola I, Zuñiga S. Biochemical aspects of coronavirus replication and virus-host interaction. Annu Rev Microbiol. 2006; 60: 211–30.
-
Li F. Structure, Function, and Evolution of Coronavirus Spike Proteins. Annu Rev Virol. 2016; 3: 237–61.
-
Khan S, Siddique R, Shereen MA, Ali A, Liu J, Bai Q, Bashir N, Xue M. The emergence of a novel coronavirus (SARS-CoV-2), their biology and therapeutic options. J Clin Microbiol [Internet]. American Society for Microbiology Journals; 2020 [cited 2020 Mar 18]; . Available from: https://jcm.asm.org/content/early/2020/03/05/JCM.00187-20
-
Fehr AR, Perlman S. Coronaviruses: An Overview of Their Replication and Pathogenesis. Methods Mol Biol Clifton NJ [Internet]. 2015 [cited 2020 Mar 18]; 1282: 1–23. Available from: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4369385/
-
Kubo H, Yamada YK, Taguchi F. Localization of neutralizing epitopes and the receptor-binding site within the amino-terminal 330 amino acids of the murine coronavirus spike protein. J Virol. 1994; 68: 5403–10.
-
Baruah V, Bose S. Immunoinformatics-aided identification of T cell and B cell epitopes in the surface glycoprotein of 2019-nCoV. J Med Virol. 2020; 92: 495–500.
-
Armstrong J, Niemann H, Smeekens S, Rottier P, Warren G. Sequence and topology of a model intracellular membrane protein, E1 glycoprotein, from a coronavirus. Nature. 1984; 308: 751–2.
-
Neuman BW, Kiss G, Kunding AH, Bhella D, Baksh MF, Connelly S, Droese B, Klaus JP, Makino S, Sawicki SG, Siddell SG, Stamou DG, Wilson IA, et al. A structural analysis of M protein in coronavirus assembly and morphology. J Struct Biol. 2011; 174: 11–22.
-
Masters PS, Kuo L, Ye R, Hurst KR, Koetzner CA, Hsue B. Genetic and molecular biological analysis of protein-protein interactions in coronavirus assembly. Adv Exp Med Biol. 2006; 581: 163–73.
-
Kuo L, Masters PS. Genetic evidence for a structural interaction between the carboxy termini of the membrane and nucleocapsid proteins of mouse hepatitis virus. J Virol. 2002; 76: 4987–99.
-
Ning Q, Lakatoo S, Liu M, Yang W, Wang Z, Phillips MJ, Levy GA. Induction of prothrombinase fgl2 by the nucleocapsid protein of virulent mouse hepatitis virus is dependent on host hepatic nuclear factor-4 alpha. J Biol Chem. 2003; 278: 15541–9.
-
Enjuanes L, Zuñiga S, Castaño-Rodriguez C, Gutierrez-Alvarez J, Canton J, Sola I. Chapter Eight - Molecular Basis of Coronavirus Virulence and Vaccine Development. In: Ziebuhr J, editor. Advances in Virus Research [Internet]. Academic Press; 2016 [cited 2020 Mar 18]. p. 245–86. Available from: http://www.sciencedirect.com/science/article/pii/S0065352716300422
-
Siu YL, Teoh KT, Lo J, Chan CM, Kien F, Escriou N, Tsao SW, Nicholls JM, Altmeyer R, Peiris JSM, Bruzzone R, Nal B. The M, E, and N structural proteins of the severe acute respiratory syndrome coronavirus are required for efficient assembly, trafficking, and release of virus-like particles. J Virol. 2008; 82: 11318–30.
-
Klausegger A, Strobl B, Regl G, Kaser A, Luytjes W, Vlasak R. Identification of a coronavirus hemagglutinin-esterase with a substrate specificity different from those of influenza C virus and bovine coronavirus. J Virol. 1999; 73: 3737–43.
-
Neuman BW, Buchmeier MJ. Chapter One - Supramolecular Architecture of the Coronavirus Particle. In: Ziebuhr J, editor. Advances in Virus Research [Internet]. Academic Press; 2016 [cited 2020 Mar 18]. p. 1–27. Available from: http://www.sciencedirect.com/science/article/pii/S0065352716300446
-
Vlasak R, Luytjes W, Spaan W, Palese P. Human and bovine coronaviruses recognize sialic acid-containing receptors similar to those of influenza C viruses. Proc Natl Acad Sci U S A. 1988; 85: 4526–9.
-
Lissenberg A, Vrolijk MM, van Vliet ALW, Langereis MA, de Groot-Mijnes JDF, Rottier PJM, de Groot RJ. Luxury at a cost? Recombinant mouse hepatitis viruses expressing the accessory hemagglutinin esterase protein display reduced fitness in vitro. J Virol. 2005; 79: 15054–63.
-
DeDiego ML, Alvarez E, Almazán F, Rejas MT, Lamirande E, Roberts A, Shieh W-J, Zaki SR, Subbarao K, Enjuanes L. A severe acute respiratory syndrome coronavirus that lacks the E gene is attenuated in vitro and in vivo. J Virol. 2007; 81: 1701–13.
-
Yount B, Roberts RS, Sims AC, Deming D, Frieman MB, Sparks J, Denison MR, Davis N, Baric RS. Severe acute respiratory syndrome coronavirus group-specific open reading frames encode nonessential functions for replication in cell cultures and mice. J Virol. 2005; 79: 14909–22.
-
de Haan CAM, Masters PS, Shen X, Weiss S, Rottier PJM. The group-specific murine coronavirus genes are not essential, but their deletion, by reverse genetics, is attenuating in the natural host. Virology. 2002; 296: 177–89.
-
Lachance C, Arbour N, Cashman NR, Talbot PJ. Involvement of aminopeptidase N (CD13) in infection of human neural cells by human coronavirus 229E. J Virol. 1998; 72: 6511–9.
-
Hofmann H, Pyrc K, van der Hoek L, Geier M, Berkhout B, Pöhlmann S. Human coronavirus NL63 employs the severe acute respiratory syndrome coronavirus receptor for cellular entry. Proc Natl Acad Sci U S A. 2005; 102: 7988–93.
-
Zaki AM, van Boheemen S, Bestebroer TM, Osterhaus ADME, Fouchier RAM. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med. 2012; 367: 1814–20.
-
Centers for Disease Control and Prevention (CDC). Severe respiratory illness associated with a novel coronavirus--Saudi Arabia and Qatar, 2012. MMWR Morb Mortal Wkly Rep. 2012; 61: 820.
-
Carstens EB. Ratification vote on taxonomic proposals to the International Committee on Taxonomy of Viruses (2009). Arch Virol. 2010; 155: 133–46.
-
Vijgen L, Keyaerts E, Moës E, Thoelen I, Wollants E, Lemey P, Vandamme A-M, Van Ranst M. Complete genomic sequence of human coronavirus OC43: molecular clock analysis suggests a relatively recent zoonotic coronavirus transmission event. J Virol. 2005; 79: 1595–604.
-
Orthocoronavirinae [Internet]. Wikipedia. 2020 [cited 2020 Mar 19]. Available from: https://it.wikipedia.org/w/index.php?title=Orthocoronavirinae&oldid=111546090
-
Snijder EJ, van der Meer Y, Zevenhoven-Dobbe J, Onderwater JJM, van der Meulen J, Koerten HK, Mommaas AM. Ultrastructure and origin of membrane vesicles associated with the severe acute respiratory syndrome coronavirus replication complex. J Virol. 2006; 80: 5927–40.
-
Stertz S, Reichelt M, Spiegel M, Kuri T, Martínez-Sobrido L, García-Sastre A, Weber F, Kochs G. The intracellular sites of early replication and budding of SARS-coronavirus. Virology. 2007; 361: 304–15.
-
Garbino J, Crespo S, Aubert J-D, Rochat T, Ninet B, Deffernez C, Wunderli W, Pache J-C, Soccal PM, Kaiser L. A prospective hospital-based study of the clinical impact of non-severe acute respiratory syndrome (Non-SARS)-related human coronavirus infection. Clin Infect Dis Off Publ Infect Dis Soc Am. 2006; 43: 1009–15.
-
Gu J, Gong E, Zhang B, Zheng J, Gao Z, Zhong Y, Zou W, Zhan J, Wang S, Xie Z, Zhuang H, Wu B, Zhong H, et al. Multiple organ infection and the pathogenesis of SARS. J Exp Med. 2005; 202: 415–24.
-
McIntosh K, Dees JH, Becker WB, Kapikian AZ, Chanock RM. Recovery in tracheal organ cultures of novel viruses from patients with respiratory disease. Proc Natl Acad Sci U S A. 1967; 57: 933–40.
-
Mihindukulasuriya KA, Wu G, St Leger J, Nordhausen RW, Wang D. Identification of a novel coronavirus from a beluga whale by using a panviral microarray. J Virol. 2008; 82: 5084–8.
-
He B, Zhang Y, Xu L, Yang W, Yang F, Feng Y, Xia L, Zhou J, Zhen W, Feng Y, Guo H, Zhang H, Tu C. Identification of diverse alphacoronaviruses and genomic characterization of a novel severe acute respiratory syndrome-like coronavirus from bats in China. J Virol. 2014; 88: 7070–82.
-
Anthony SJ, Johnson CK, Greig DJ, Kramer S, Che X, Wells H, Hicks AL, Joly DO, Wolfe ND, Daszak P, Karesh W, Lipkin WI, Morse SS, et al. Global patterns in coronavirus diversity. Virus Evol. 2017; 3: vex012.
-
Chinese SARS Molecular Epidemiology Consortium. Molecular evolution of the SARS coronavirus during the course of the SARS epidemic in China. Science. 2004; 303: 1666–9.
-
Song H-D, Tu C-C, Zhang G-W, Wang S-Y, Zheng K, Lei L-C, Chen Q-X, Gao Y-W, Zhou H-Q, Xiang H, Zheng H-J, Chern S-WW, Cheng F, et al. Cross-host evolution of severe acute respiratory syndrome coronavirus in palm civet and human. Proc Natl Acad Sci U S A. 2005; 102: 2430–5.
-
Li W, Zhang C, Sui J, Kuhn JH, Moore MJ, Luo S, Wong S-K, Huang I-C, Xu K, Vasilieva N, Murakami A, He Y, Marasco WA, et al. Receptor and viral determinants of SARS-coronavirus adaptation to human ACE2. EMBO J. 2005; 24: 1634–43.
-
Sheahan T, Rockx B, Donaldson E, Corti D, Baric R. Pathways of cross-species transmission of synthetically reconstructed zoonotic severe acute respiratory syndrome coronavirus. J Virol. 2008; 82: 8721–32.
-
Li W, Shi Z, Yu M, Ren W, Smith C, Epstein JH, Wang H, Crameri G, Hu Z, Zhang H, Zhang J, McEachern J, Field H, et al. Bats are natural reservoirs of SARS-like coronaviruses. Science. 2005; 310: 676–9.
-
Lau SKP, Woo PCY, Li KSM, Huang Y, Tsoi H-W, Wong BHL, Wong SSY, Leung S-Y, Chan K-H, Yuen K-Y. Severe acute respiratory syndrome coronavirus-like virus in Chinese horseshoe bats. Proc Natl Acad Sci U S A. 2005; 102: 14040–5.
-
Leung DTM, van Maren WWC, Chan FKL, Chan WS, Lo AWI, Ma CH, Tam FCH, To KF, Chan PKS, Sung JJY, Lim PL. Extremely low exposure of a community to severe acute respiratory syndrome coronavirus: false seropositivity due to use of bacterially derived antigens. J Virol. 2006; 80: 8920–8.
-
Killerby ME, Biggs HM, Haynes A, Dahl RM, Mustaquim D, Gerber SI, Watson JT. Human coronavirus circulation in the United States 2014-2017. J Clin Virol Off Publ Pan Am Soc Clin Virol. 2018; 101: 52–6.
-
Zeng Z-Q, Chen D-H, Tan W-P, Qiu S-Y, Xu D, Liang H-X, Chen M-X, Li X, Lin Z-S, Liu W-K, Zhou R. Epidemiology and clinical characteristics of human coronaviruses OC43, 229E, NL63, and HKU1: a study of hospitalized children with acute respiratory tract infection in Guangzhou, China. Eur J Clin Microbiol Infect Dis Off Publ Eur Soc Clin Microbiol. 2018; 37: 363–9.
-
Heimdal I, Moe N, Krokstad S, Christensen A, Skanke LH, Nordbø SA, Døllner H. Human Coronavirus in Hospitalized Children With Respiratory Tract Infections: A 9-Year Population-Based Study From Norway. J Infect Dis. 2019; 219: 1198–206.
-
Friedman N, Alter H, Hindiyeh M, Mendelson E, Shemer Avni Y, Mandelboim M. Human Coronavirus Infections in Israel: Epidemiology, Clinical Symptoms and Summer Seasonality of HCoV-HKU1. Viruses. 2018; 10.
-
McIntosh K, Chao RK, Krause HE, Wasil R, Mocega HE, Mufson MA. Coronavirus infection in acute lower respiratory tract disease of infants. J Infect Dis. 1974; 130: 502–7.
-
McIntosh K, Kapikian AZ, Turner HC, Hartley JW, Parrott RH, Chanock RM. Seroepidemiologic studies of coronavirus infection in adults and children. Am J Epidemiol. 1970; 91: 585–92.
-
Soonnarong R, Thongpan I, Payungporn S, Vuthitanachot C, Vuthitanachot V, Vichiwattana P, Vongpunsawad S, Poovorawan Y. Molecular epidemiology and characterization of human coronavirus in Thailand, 2012-2013. SpringerPlus. 2016; 5: 1420.
-
Nickbakhsh S, Thorburn F, VON Wissmann B, McMENAMIN J, Gunson RN, Murcia PR. Extensive multiplex PCR diagnostics reveal new insights into the epidemiology of viral respiratory infections. Epidemiol Infect. 2016; 144: 2064–76.
-
Reed SE. The behaviour of recent isolates of human respiratory coronavirus in vitro and in volunteers: evidence of heterogeneity among 229E-related strains. J Med Virol. 1984; 13: 179–92.
-
Gagneur A, Vallet S, Talbot PJ, Legrand-Quillien M-C, Picard B, Payan C, Sizun J. Outbreaks of human coronavirus in a pediatric and neonatal intensive care unit. Eur J Pediatr. 2008; 167: 1427–34.
-
Falsey AR, Dallal GE, Formica MA, Andolina GG, Hamer DH, Leka LL, Meydani SN. Long-term care facilities: a cornucopia of viral pathogens. J Am Geriatr Soc. 2008; 56: 1281–5.
-
Bradburne AF, Somerset BA. Coronative antibody tires in sera of healthy adults and experimentally infected volunteers. J Hyg (Lond). 1972; 70: 235–44.
-
McIntosh K, Ellis EF, Hoffman LS, Lybass TG, Eller JJ, Fulginiti VA. Association of Viral and Bacterial Respiratory Infection with Exacerbations of Wheezing in Young Asthmatic Children. CHEST [Internet]. Elsevier; 1973 [cited 2020 Mar 18]; 63: 43S. Available from: https://journal.chestnet.org/article/S0012-3692(15)48593-9/abstract
-
Fouchier RAM, Hartwig NG, Bestebroer TM, Niemeyer B, de Jong JC, Simon JH, Osterhaus ADME. A previously undescribed coronavirus associated with respiratory disease in humans. Proc Natl Acad Sci U S A. 2004; 101: 6212–6.
-
van der Hoek L, Pyrc K, Jebbink MF, Vermeulen-Oost W, Berkhout RJM, Wolthers KC, Wertheim-van Dillen PME, Kaandorp J, Spaargaren J, Berkhout B. Identification of a new human coronavirus. Nat Med. 2004; 10: 368–73.
-
Woo PCY, Lau SKP, Chu C, Chan K, Tsoi H, Huang Y, Wong BHL, Poon RWS, Cai JJ, Luk W, Poon LLM, Wong SSY, Guan Y, et al. Characterization and complete genome sequence of a novel coronavirus, coronavirus HKU1, from patients with pneumonia. J Virol. 2005; 79: 884–95.
-
Pyrc K, Dijkman R, Deng L, Jebbink MF, Ross HA, Berkhout B, van der Hoek L. Mosaic structure of human coronavirus NL63, one thousand years of evolution. J Mol Biol. 2006; 364: 964–73.
-
Chavanet P. Viral Upper Respiratory Tract Infection and Otitis Media Complication in Young Children. Clin Infect Dis [Internet]. Oxford Academic; 2008 [cited 2020 Mar 18]; 46: 824–824. Available from: https://academic.oup.com/cid/article/46/6/824/347801
-
Chonmaitree T, Revai K, Grady JJ, Clos A, Patel JA, Nair S, Fan J, Henrickson KJ. Viral upper respiratory tract infection and otitis media complication in young children. Clin Infect Dis Off Publ Infect Dis Soc Am. 2008; 46: 815–23.
-
Prill MM, Iwane MK, Edwards KM, Williams JV, Weinberg GA, Staat MA, Willby MJ, Talbot HK, Hall CB, Szilagyi PG, Griffin MR, Curns AT, Erdman DD, et al. Human coronavirus in young children hospitalized for acute respiratory illness and asymptomatic controls. Pediatr Infect Dis J. 2012; 31: 235–40.
-
Self WH, Williams DJ, Zhu Y, Ampofo K, Pavia AT, Chappell JD, Hymas WC, Stockmann C, Bramley AM, Schneider E, Erdman D, Finelli L, Jain S, et al. Respiratory Viral Detection in Children and Adults: Comparing Asymptomatic Controls and Patients With Community-Acquired Pneumonia. J Infect Dis. 2016; 213: 584–91.
-
Ieven M, Coenen S, Loens K, Lammens C, Coenjaerts F, Vanderstraeten A, Henriques-Normark B, Crook D, Huygen K, Butler CC, Verheij TJM, Little P, Zlateva K, et al. Aetiology of lower respiratory tract infection in adults in primary care: a prospective study in 11 European countries. Clin Microbiol Infect Off Publ Eur Soc Clin Microbiol Infect Dis. 2018; 24: 1158–63.
-
Lieberman D, Shimoni A, Shemer-Avni Y, Keren-Naos A, Shtainberg R, Lieberman D. Respiratory viruses in adults with community-acquired pneumonia. Chest. 2010; 138: 811–6.
-
Wansaula Z, Olsen SJ, Casal MG, Golenko C, Erhart LM, Kammerer P, Whitfield N, McCotter OZ. Surveillance for severe acute respiratory infections in Southern Arizona, 2010-2014. Influenza Other Respir Viruses. 2016; 10: 161–9.
-
Nicholson KG, Kent J, Hammersley V, Cancio E. Acute viral infections of upper respiratory tract in elderly people living in the community: comparative, prospective, population based study of disease burden. BMJ. 1997; 315: 1060–4.
-
Graat JM, Schouten EG, Heijnen M-LA, Kok FJ, Pallast EGM, de Greeff SC, Dorigo-Zetsma JW. A prospective, community-based study on virologic assessment among elderly people with and without symptoms of acute respiratory infection. J Clin Epidemiol. 2003; 56: 1218–23.
-
Falsey AR, McCann RM, Hall WJ, Criddle MM, Formica MA, Wycoff D, Kolassa JE. The “common cold” in frail older persons: impact of rhinovirus and coronavirus in a senior daycare center. J Am Geriatr Soc. 1997; 45: 706–11.
-
Patrick DM, Petric M, Skowronski DM, Guasparini R, Booth TF, Krajden M, McGeer P, Bastien N, Gustafson L, Dubord J, Macdonald D, David ST, Srour LF, et al. An Outbreak of Human Coronavirus OC43 Infection and Serological Cross-reactivity with SARS Coronavirus. Can J Infect Dis Med Microbiol J Can Mal Infect Microbiol Medicale. 2006; 17: 330–6.
-
Galante O, Avni YS, Fuchs L, Ferster OA, Almog Y. Coronavirus NL63-induced Adult Respiratory Distress Syndrome. Am J Respir Crit Care Med. 2016; 193: 100–1.
-
Nicholson KG, Kent J, Ireland DC. Respiratory viruses and exacerbations of asthma in adults. BMJ. 1993; 307: 982–6.
-
McIntosh K, Ellis EF, Hoffman LS, Lybass TG, Eller JJ, Fulginiti VA. The association of viral and bacterial respiratory infections with exacerbations of wheezing in young asthmatic children. J Pediatr. 1973; 82: 578–90.
-
Sizun J, Soupre D, Legrand MC, Giroux JD, Rubio S, Cauvin JM, Chastel C, Alix D, de Parscau L. Neonatal nosocomial respiratory infection with coronavirus: a prospective study in a neonatal intensive care unit. Acta Paediatr Oslo Nor 1992. 1995; 84: 617–20.
-
van der Hoek L, Sure K, Ihorst G, Stang A, Pyrc K, Jebbink MF, Petersen G, Forster J, Berkhout B, Uberla K. Croup is associated with the novel coronavirus NL63. PLoS Med. 2005; 2: e240.
-
Pene F, Merlat A, Vabret A, Rozenberg F, Buzyn A, Dreyfus F, Cariou A, Freymuth F, Lebon P. Coronavirus 229E-related pneumonia in immunocompromised patients. Clin Infect Dis Off Publ Infect Dis Soc Am. 2003; 37: 929–32.
-
Szczawinska-Poplonyk A, Jonczyk-Potoczna K, Breborowicz A, Bartkowska-Sniatkowska A, Figlerowicz M. Fatal respiratory distress syndrome due to coronavirus infection in a child with severe combined immunodeficiency. Influenza Other Respir Viruses. 2013; 7: 634–6.
-
Simon A, Völz S, Fleischhack G, Tillman R, Müller A, Bode U, Schildgen O. Human coronavirus OC43 pneumonia in a pediatric cancer patient with down syndrome and acute lymphoblastic leukemia. J Pediatr Hematol Oncol. 2007; 29: 432–4.
-
Ogimi C, Waghmare AA, Kuypers JM, Xie H, Yeung CC, Leisenring WM, Seo S, Choi S-M, Jerome KR, Englund JA, Boeckh M. Clinical Significance of Human Coronavirus in Bronchoalveolar Lavage Samples From Hematopoietic Cell Transplant Recipients and Patients With Hematologic Malignancies. Clin Infect Dis Off Publ Infect Dis Soc Am. 2017; 64: 1532–9.
-
Nunes MC, Kuschner Z, Rabede Z, Madimabe R, Van Niekerk N, Moloi J, Kuwanda L, Rossen JW, Klugman KP, Adrian PV, Madhi SA. Clinical epidemiology of bocavirus, rhinovirus, two polyomaviruses and four coronaviruses in HIV-infected and HIV-uninfected South African children. PloS One. 2014; 9: e86448.
-
Garbino J, Inoubli S, Mossdorf E, Weber R, Tamm M, Soccal P, Aubert J-D, Bridevaux P-O, Tapparel C, Kaiser L, Swiss HIV Cohort Study. Respiratory viruses in HIV-infected patients with suspected respiratory opportunistic infection. AIDS Lond Engl. 2008; 22: 701–5.
-
Kumar D, Husain S, Chen MH, Moussa G, Himsworth D, Manuel O, Studer S, Pakstis D, McCurry K, Doucette K, Pilewski J, Janeczko R, Humar A. A prospective molecular surveillance study evaluating the clinical impact of community-acquired respiratory viruses in lung transplant recipients. Transplantation. 2010; 89: 1028–33.
-
Chany C, Moscovici O, Lebon P, Rousset S. Association of coronavirus infection with neonatal necrotizing enterocolitis. Pediatrics. 1982; 69: 209–14.
-
Gerna G, Passarani N, Battaglia M, Rondanelli EG. Human enteric coronaviruses: antigenic relatedness to human coronavirus OC43 and possible etiologic role in viral gastroenteritis. J Infect Dis. 1985; 151: 796–803.
-
Jevšnik M, Steyer A, Zrim T, Pokorn M, Mrvič T, Grosek Š, Strle F, Lusa L, Petrovec M. Detection of human coronaviruses in simultaneously collected stool samples and nasopharyngeal swabs from hospitalized children with acute gastroenteritis. Virol J. 2013; 10: 46.
-
Risku M, Lappalainen S, Räsänen S, Vesikari T. Detection of human coronaviruses in children with acute gastroenteritis. J Clin Virol Off Publ Pan Am Soc Clin Virol. 2010; 48: 27–30.
-
Esper F, Ou Z, Huang YT. Human coronaviruses are uncommon in patients with gastrointestinal illness. J Clin Virol Off Publ Pan Am Soc Clin Virol. 2010; 48: 131–3.
-
Minodier L, Masse S, Capai L, Blanchon T, Ceccaldi P-E, van der Werf S, Hanslik T, Charrel R, Falchi A. Clinical and virological factors associated with gastrointestinal symptoms in patients with acute respiratory infection: a two-year prospective study in general practice medicine. BMC Infect Dis. 2017; 17: 729.
-
Arbour N, Ekandé S, Côté G, Lachance C, Chagnon F, Tardieu M, Cashman NR, Talbot PJ. Persistent infection of human oligodendrocytic and neuroglial cell lines by human coronavirus 229E. J Virol. 1999; 73: 3326–37.
-
Jacomy H, Talbot PJ. Vacuolating encephalitis in mice infected by human coronavirus OC43. Virology. 2003; 315: 20–33.
-
Yeh EA, Collins A, Cohen ME, Duffner PK, Faden H. Detection of coronavirus in the central nervous system of a child with acute disseminated encephalomyelitis. Pediatrics. 2004; 113: e73-76.
-
Morfopoulou S, Brown JR, Davies EG, Anderson G, Virasami A, Qasim W, Chong WK, Hubank M, Plagnol V, Desforges M, Jacques TS, Talbot PJ, Breuer J. Human Coronavirus OC43 Associated with Fatal Encephalitis. N Engl J Med. 2016; 375: 497–8.
-
Houtman JJ, Fleming JO. Pathogenesis of mouse hepatitis virus-induced demyelination. J Neurovirol. 1996; 2: 361–76.
-
Boucher A, Desforges M, Duquette P, Talbot PJ. Long-term human coronavirus-myelin cross-reactive T-cell clones derived from multiple sclerosis patients. Clin Immunol Orlando Fla. 2007; 123: 258–67.
-
Arbour N, Day R, Newcombe J, Talbot PJ. Neuroinvasion by human respiratory coronaviruses. J Virol. 2000; 74: 8913–21.
-
Desforges M, Le Coupanec A, Dubeau P, Bourgouin A, Lajoie L, Dubé M, Talbot PJ. Human Coronaviruses and Other Respiratory Viruses: Underestimated Opportunistic Pathogens of the Central Nervous System? Viruses. 2019; 12.
-
Esper F, Shapiro ED, Weibel C, Ferguson D, Landry ML, Kahn JS. Association between a novel human coronavirus and Kawasaki disease. J Infect Dis. 2005; 191: 499–502.
-
Chang L-Y, Chiang B-L, Kao C-L, Wu M-H, Chen P-J, Berkhout B, Yang H-C, Huang L-M, Kawasaki Disease Research Group. Lack of association between infection with a novel human coronavirus (HCoV), HCoV-NH, and Kawasaki disease in Taiwan. J Infect Dis. 2006; 193: 283–6.
-
Dominguez SR, Anderson MS, Glodé MP, Robinson CC, Holmes KV. Blinded case-control study of the relationship between human coronavirus NL63 and Kawasaki syndrome. J Infect Dis. 2006; 194: 1697–701.
-
Rothan HA, Byrareddy SN. The epidemiology and pathogenesis of coronavirus disease (COVID-19) outbreak. J Autoimmun. 2020; : 102433.
-
Liu L, Wang T, Lu J. The prevalence, origin, and prevention of six human coronaviruses. Virol Sin. 2016; 31: 94–9.
-
Gerna G, Percivalle E, Sarasini A, Campanini G, Piralla A, Rovida F, Genini E, Marchi A, Baldanti F. Human respiratory coronavirus HKU1 versus other coronavirus infections in Italian hospitalised patients. J Clin Virol Off Publ Pan Am Soc Clin Virol. 2007; 38: 244–50.
-
Sizun J, Arbour N, Talbot PJ. Comparison of immunofluorescence with monoclonal antibodies and RT-PCR for the detection of human coronaviruses 229E and OC43 in cell culture. J Virol Methods. 1998; 72: 145–52.
-
Zlateva KT, Coenjaerts FEJ, Crusio KM, Lammens C, Leus F, Viveen M, Ieven M, Spaan WJM, Claas ECJ, Gorbalenya AE. No novel coronaviruses identified in a large collection of human nasopharyngeal specimens using family-wide CODEHOP-based primers. Arch Virol. 2013; 158: 251–5.
-
Keyaerts E, Vijgen L, Maes P, Neyts J, Van Ranst M. In vitro inhibition of severe acute respiratory syndrome coronavirus by chloroquine. Biochem Biophys Res Commun. 2004; 323: 264–8.
-
Keyaerts E, Li S, Vijgen L, Rysman E, Verbeeck J, Van Ranst M, Maes P. Antiviral activity of chloroquine against human coronavirus OC43 infection in newborn mice. Antimicrob Agents Chemother. 2009; 53: 3416–21.
-
Ryu S, Chun BC. An interim review of the epidemiological characteristics of 2019 novel coronavirus. Epidemiol Health [Internet]. 2020 [cited 2020 Mar 18]; 42. Available from: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7011107/
-
Otter JA, Donskey C, Yezli S, Douthwaite S, Goldenberg SD, Weber DJ. Transmission of SARS and MERS coronaviruses and influenza virus in healthcare settings: the possible role of dry surface contamination. J Hosp Infect. 2016; 92: 235–50.
-
Geller C, Varbanov M, Duval RE. Human coronaviruses: insights into environmental resistance and its influence on the development of new antiseptic strategies. Viruses. 2012; 4: 3044–68.
-
Sattar SA, Springthorpe VS, Karim Y, Loro P. Chemical disinfection of non-porous inanimate surfaces experimentally contaminated with four human pathogenic viruses. Epidemiol Infect. 1989; 102: 493–505.
-
Hulkower RL, Casanova LM, Rutala WA, Weber DJ, Sobsey MD. Inactivation of surrogate coronaviruses on hard surfaces by health care germicides. Am J Infect Control. 2011; 39: 401–7.
-
Dellanno C, Vega Q, Boesenberg D. The antiviral action of common household disinfectants and antiseptics against murine hepatitis virus, a potential surrogate for SARS coronavirus. Am J Infect Control. 2009; 37: 649–52.
-
Cao J, Forrest JC, Zhang X. A screen of the NIH Clinical Collection small molecule library identifies potential anti-coronavirus drugs. Antiviral Res. 2015; 114: 1–10.
-
Vennema H, de Groot RJ, Harbour DA, Dalderup M, Gruffydd-Jones T, Horzinek MC, Spaan WJ. Early death after feline infectious peritonitis virus challenge due to recombinant vaccinia virus immunization. J Virol. 1990; 64: 1407–9.
